Saiba como usar ferramentas de OCR, Apache Spark e outros componentes do Apache Hadoop para processar imagens PDF em escala.
As tecnologias de reconhecimento óptico de caracteres (OCR) avançaram significativamente nos últimos 20 anos. No entanto, durante esse período, houve pouco ou nenhum esforço para casar o OCR com arquiteturas distribuídas, como o Apache Hadoop, para processar um grande número de imagens quase em tempo real.
Nesta postagem, você aprenderá a usar ferramentas padrão de código aberto junto com componentes do Hadoop, como Apache Spark, Apache Solr e Apache HBase, para fazer exatamente isso para um caso de uso de informações de dispositivos médicos. Especificamente, você usará um conjunto de dados público para converter texto narrativo em campos pesquisáveis.
Embora este exemplo se concentre em informações de dispositivos médicos, ele pode ser aplicado em muitos outros cenários em que o processamento e a persistência de imagens são necessários. As companhias de seguros, por exemplo, podem tornar todos os seus documentos digitalizados em arquivos de sinistros pesquisáveis para uma melhor resolução de sinistros. Da mesma forma, o departamento de cadeia de suprimentos em uma fábrica pode escanear todas as fichas técnicas de fornecedores de peças e torná-las pesquisáveis por analistas.
Caso de uso:registro de dispositivo médico
Nos últimos anos, houve uma enxurrada de mudanças no campo do registro eletrônico de medicamentos. O padrão ISO IDMP (Identificação de produtos médicos) é um desses formatos de mensagem para registro de produtos e das substâncias neles contidas, com o ID do Medicamento, o ID da Embalagem e o ID do Lote sendo usados para rastrear os produtos nos casos de experiências adversas, importação, falsificação e outras questões de farmacovigilância. A norma exige que não apenas os novos produtos sejam registrados, mas que o arquivamento mais antigo/arquivado de todos os produtos aos quais o público possa estar exposto também seja fornecido em formato eletrônico.
Para cumprir os padrões IDMP em diferentes empresas, as empresas devem ser capazes de extrair e processar dados de várias fontes de dados, como RDBMS e, em alguns casos, folhas de dados de produtos herdados. Embora seja bem conhecido como ingerir dados do RDBMS por meio de tecnologias como Apache Sqoop, o processamento de documentos legados requer um pouco mais de trabalho. Na maioria das vezes, os documentos precisam ser ingeridos e o texto relevante precisa ser extraído programaticamente em escala usando as tecnologias de OCR existentes.
Conjunto de dados
Usaremos um conjunto de dados da FDA que contém todos os registros 510(k) já enviados por fabricantes de dispositivos médicos desde 1976. A Seção 510(k) da Lei de Alimentos, Medicamentos e Cosméticos exige que os fabricantes de dispositivos que devem se registrar notifiquem FDA de sua intenção de comercializar um dispositivo médico com pelo menos 90 dias de antecedência.
Este conjunto de dados é útil por vários motivos neste caso:
- Os dados são gratuitos e de domínio público.
- Os dados estão de acordo com o regulamento europeu, que entra em vigor em julho de 2016 (onde os fabricantes devem cumprir os novos padrões de dados). Os preenchimentos da FDA têm informações importantes relevantes para obter uma visão completa do IDMP.
- O formato dos documentos (PDF) nos permite demonstrar técnicas de OCR simples e eficazes ao lidar com documentos de vários formatos.
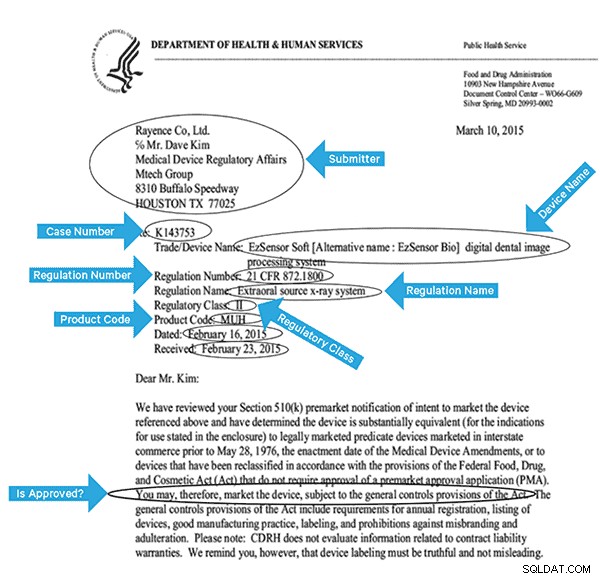
Para indexar efetivamente esses dados, precisaremos extrair alguns campos das imagens. Abaixo está um documento de amostra, com os campos potenciais que podem ser extraídos.
Arquitetura de alto nível
Para este caso de uso, os PDFs são armazenados em HDFS e processados usando bibliotecas Spark e OCR. (A etapa de ingestão está fora do escopo desta postagem, mas pode ser tão simples quanto executar
hdfs -dfs -put ou usando uma interface webhdfs.) O Spark permite o uso de código quase idêntico em um aplicativo Spark Streaming para streaming quase em tempo real, e o HBase é um meio de armazenamento perfeito para acesso aleatório de baixa latência - e é adequado para armazenar imagens, com a nova funcionalidade MOB, para inicializar. Cloudera Search (que é construído em cima do Apache Solr) é a única solução de pesquisa que se integra nativamente com o HBase, permitindo que você crie índices secundários. 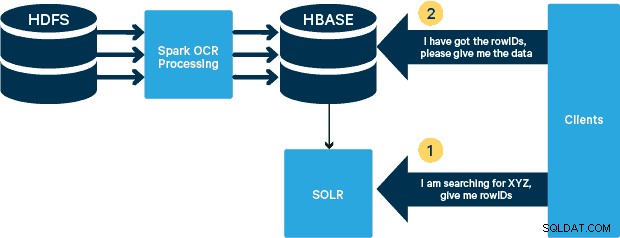
Configurando a Tabela de Dispositivos Médicos no HBase
Manteremos o esquema para nosso caso de uso simples. O rowID será o nome do arquivo e haverá duas famílias de colunas:“info” e “obj”. A família de colunas “info” conterá todos os campos que extraímos das imagens. A família de colunas “obj” conterá os bytes do objeto binário real, neste caso PDF. O nome da tabela no nosso caso será “mdds”.
Aproveitaremos a funcionalidade HBase MOB (objeto médio) introduzida no HBASE-11339. Para configurar o HBase para lidar com o MOB, algumas etapas extras são necessárias, mas, convenientemente, as instruções podem ser encontradas neste link.
Há muitas maneiras de criar a tabela no HBase programaticamente (API Java, API REST ou um método semelhante). Aqui usaremos o shell HBase para criar a tabela “mdds” (intencionalmente usando um nome de família de colunas descritivo para facilitar o acompanhamento). Queremos que a família de colunas “info” seja replicada para o Solr, mas não os dados MOB.
O comando abaixo criará a tabela e habilitará a replicação em um grupo de colunas chamado “info”. É crucial especificar a opção
REPLICATION_SCOPE => '1' , caso contrário, o HBase Lily Indexer não receberá nenhuma atualização do HBase. Queremos usar o caminho MOB no HBase para objetos maiores que 10 MB. Para isso, também criamos outra família de colunas, chamada “obj”, usando os seguintes parâmetros para MOBs:IS_MOB => verdadeiro, MOB_THRESHOLD => 10240000
O
IS_MOB O parâmetro especifica se este grupo de colunas pode armazenar MOBs, enquanto MOB_THRESHOLD especifica depois de quão grande o objeto deve ser para que seja considerado um MOB. Então, vamos criar a tabela:criar 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000}
Para confirmar que a tabela foi criada corretamente, execute o seguinte comando no shell do HBase:
hbase(main):001:0> descreve 'mdds'Table mdds is ENABLEDmddsCOLUMN FAMILIES DESCRIPTION{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1' , VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'FOREVER', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}2 linhas em 0,3440 segundos Processando imagens digitalizadas com o Tesseract
O OCR percorreu um longo caminho em termos de lidar com variações de fonte, ruído de imagem e problemas de alinhamento. Aqui, usaremos o mecanismo de OCR de código aberto Tesseract, que foi originalmente desenvolvido como software proprietário nos laboratórios da HP. O desenvolvimento do Tesseract foi lançado como um software de código aberto e patrocinado pelo Google desde 2006.
Tesseract é uma biblioteca de software altamente portátil. Ele usa a biblioteca de processamento de imagem Leptonica para gerar uma imagem binária fazendo um limiar adaptativo em uma imagem cinza ou colorida.
O processamento segue um pipeline tradicional passo a passo. A seguir está o fluxo aproximado de etapas:
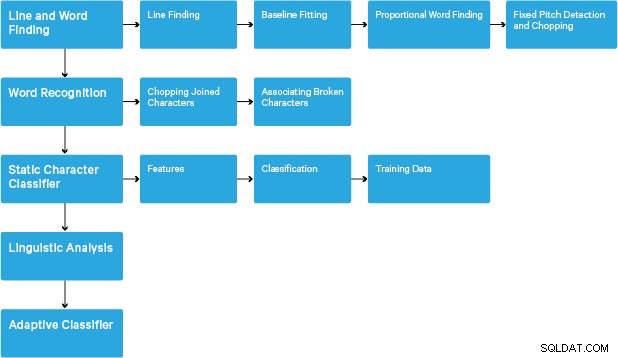
O processamento começa com uma análise de componentes conectados, que resulta no armazenamento dos componentes encontrados. Esta etapa ajuda na inspeção do aninhamento de contornos e do número de contornos filho e neto.
Nesta fase, os contornos são reunidos, puramente por aninhamento, em Binary Large Objects (BLOBs). Os BLOBs são organizados em linhas de texto e as linhas e regiões são analisadas para pitch fixo ou texto proporcional. As linhas de texto são divididas em palavras de forma diferente de acordo com o tipo de espaçamento entre caracteres. O texto de pitch fixo é cortado imediatamente por células de caracteres. O texto proporcional é dividido em palavras usando espaços definidos e espaços difusos.
O reconhecimento então prossegue como um processo de duas etapas. Na primeira passagem, é feita uma tentativa de reconhecer cada palavra por vez. Cada palavra que é satisfatória é passada para um classificador adaptativo como dados de treinamento. O classificador adaptável tem a chance de reconhecer com mais precisão o texto mais abaixo na página. Como o classificador adaptativo pode ter aprendido algo útil tarde demais para fazer uma contribuição perto do topo da página, uma segunda passagem é executada na página, na qual as palavras que não foram reconhecidas suficientemente bem são reconhecidas novamente. Uma fase final resolve os espaços difusos e verifica hipóteses alternativas para a altura x para localizar o texto em versalete.
O Tesseract em sua forma atual é totalmente compatível com Unicode e treinado para vários idiomas. Com base em nossa pesquisa, é uma das bibliotecas de código aberto mais precisas disponíveis para OCR. Como mencionado anteriormente, o Tesseract usa Leptonica. Também usamos o Ghostscript para dividir os arquivos PDF em imagens. (Você pode dividir no formato de compactação de imagem de sua escolha; escolhemos PNG.) Essas três bibliotecas são escritas em C++ e, para invocá-las de programas Java/Scala, precisamos usar implementações de interfaces nativas Java correspondentes. Em nosso trabalho, usamos as ligações JNI de JavaPresets. (As instruções de compilação podem ser encontradas abaixo.) Usamos Scala para escrever o driver Spark.
val renderer :SimpleRenderer =novo SimpleRenderer( )renderer.setResolution( 300 )val images:List[Image] =renderer.render( document )
Leptonica lê as imagens divididas da etapa anterior.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix:PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacidade( ))
Em seguida, usamos as chamadas da API do Tesseract para extrair o texto. Assumimos que os documentos estão em inglês aqui, portanto, o segundo parâmetro para o método Init é “eng”.
val api:TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
Após o processamento das imagens, extraímos alguns campos do texto e os enviamos para o HBase.
def populateHbase ( fileName:String, lines:String, pdf:org.apache.spark.input.PortableDataStream) :Unit ={ /** Configure e abra uma conexão HBase */ val mddsTbl =_conn.getTable( TableName. valueOf( "mdds" )); val cf ="info" val put =new Put( Bytes.toBytes( fileName )) /** * Extrair campos aqui usando Regexes * Criar objetos Put e enviar para HBase */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe:(\w\d\d\d\d\d\d).*"" ".r …….. lines match { case aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( addr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "não correspondeu a um regex" ) } ……. lines.split("\n").foreach { val regNumRegex ="""Número do Regulamento:\s+(.+)""".r val regNameRegex ="""Nome do Regulamento:\s+(.+)""" .r …….. ……. _ match { case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "" ) } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "texto" ), Bytes.toBytes( linhas )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( put ) …….}
Se você observar atentamente o código acima, logo antes de enviarmos o objeto Put para o HBase, inserimos os bytes do PDF bruto na família de colunas “obj” da tabela. Usamos o HBase como camada de armazenamento para os campos extraídos, bem como para a imagem bruta. Isso torna rápido e conveniente para o aplicativo extrair a imagem original, se necessário. O código completo pode ser encontrado aqui. (Vale a pena notar que, embora tenhamos usado APIs HBase padrão para criar objetos Put para HBase, em um sistema de produção real, seria sensato considerar o uso de APIs SparkOnHBase, que permitem atualizações em lote para HBase a partir de Spark RDDs.)
Canal de Execução
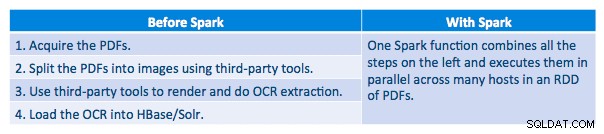
Conseguimos processar cada PDF em uma estrutura serial. Para dimensionar o processamento, optamos por processar esses PDFs de forma distribuída usando o Spark. O gráfico a seguir demonstra como combinamos diferentes estágios desse processamento para transformar o fluxo de trabalho em uma simples chamada de macro do Spark e obter os dados carregados no HBase.
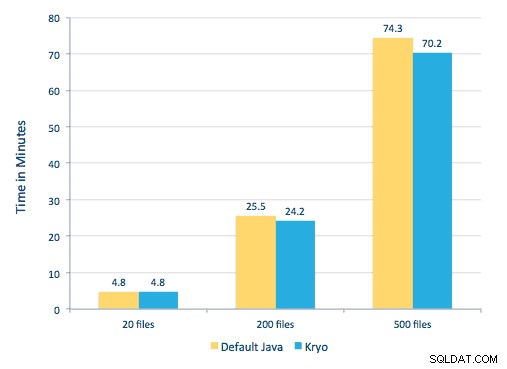
Também tentamos fazer uma comparação entre os métodos de serialização, mas, com nosso conjunto de dados, não vimos diferença significativa no desempenho.
Configuração do ambiente
Hardware usado:cluster de cinco nós com memória de 15 GB, 4 vCPUs e SSD de 2 x 40 GB
Como estávamos usando bibliotecas C++ para processamento, usamos as ligações JNI que podem ser encontradas aqui.
Crie as ligações JNI para Tesseract e Leptonica a partir de predefinições javaCPP:
-
- Em todos os nós:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel
git clone https://github.com/bytedeco/javacpp-presets.git cd javacpp-presets - Crie o Leptonica.
cd leptonica./cppbuild.sh instale o leptonicacd cppbuild/linux-x86_64/leptonica-1.72/LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&sudo make installcd ../../../mvn clean installcd ..
- Crie o Tesseract.
cd tesseract./cppbuild.sh install tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ../ ../../mvn clean installcd ..
- Crie predefinições javaCPP.
mvn clean install --projects leptonica,tesseract
Usamos o Ghostscript para extrair as imagens dos PDFs. As instruções para construir o Ghostscript, correspondentes às versões do Tesseract e Leptonica usadas aqui, são as seguintes. (Certifique-se de que o Ghostscript não esteja instalado no sistema por meio do gerenciador de pacotes.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo make &&make soinstall &&install -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps(Dependendo do seu ldpath configuração, você pode ter que fazer):sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Certifique-se de que todas as bibliotecas necessárias estejam no caminho de classe. Colocamos todos os jars relevantes em um diretório chamado lib. A vírgula é importante abaixo:
$ para i em `ls lib/*`; exporte MY_JARS=./$i,$MY_JARS; dotesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Invocamos o programa Spark da seguinte forma. Precisamos especificar o extraLibraryPath para bibliotecas nativas do Ghostscript; o outro conf é necessário para o Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase /lib/htrace-core-3.1.0-incubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Criando uma coleção Solr
O Solr integra-se perfeitamente com o HBase através do Lily HBase Indexer. Para entender como é feita a integração do Lily Indexer com o HBase, você pode revisar nossa postagem anterior na seção “Entendendo a replicação do HBase e o Lily HBase Indexer”.
Abaixo, descrevemos as etapas que precisam ser executadas para criar os índices:
- Gere um arquivo de configuração schema.xml de amostra:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg
- Edite o arquivo schema.xml em
$HOME/solrcfg , especificando os campos que precisamos para nossa coleção. O arquivo completo pode ser encontrado aqui.
- Faça upload das configurações do Solr para o ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg
- Gere a coleção Solr com 2 fragmentos (-s 2) e 2 réplicas (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
No comando acima criamos uma coleção Solr com dois parâmetros de shards (-s 2) e duas réplicas (-r 2). Os parâmetros foram suficientes para o nosso corpus, mas em uma implantação real seria necessário definir o número com base em outras considerações fora do nosso escopo de discussão aqui.
Registrando o Indexador
Esta etapa é necessária para adicionar e configurar o indexador e a replicação do HBase. O comando abaixo atualizará o ZooKeeper e adicionará mdds_indexer como um ponto de replicação para o HBase. Ele também inserirá configurações no ZooKeeper, que o Lily HBase Indexer usará para apontar para a coleção correta no Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumentos:
-n mdds_indexer– especifica o nome do indexador que será registrado no ZooKeeper-c indexer-config.xml– arquivo de configuração que especificará o comportamento do indexador-cp solr.zk=localhost:2181/solr– especifica a localização da configuração do ZooKeeper e Solr. Isso deve ser atualizado com a localização específica do ambiente do ZooKeeper.-cp solr.collection=mdds_collection– especifica qual coleção atualizar. Lembre-se da etapa de configuração do Solr em que criamos a coleção1.
O
index-config.xml file é relativamente simples neste caso; tudo o que ele faz é especificar para o indexador qual tabela examinar, a classe que será usada como mapeador (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ) e o local do arquivo de configuração Morphline. Por padrão, o tipo de mapeamento é definido como linha , caso em que o documento Solr se torna a linha completa. Param name="morphlineFile" especifica o local do arquivo de configuração Morphlines. O local pode ser um caminho absoluto do seu arquivo Morphlines, mas como você está usando o Cloudera Manager, especifique o caminho relativo como morphlines.conf. O conteúdo do arquivo de configuração do hbase-indexer pode ser encontrado aqui.
Configurando e iniciando o Lily HBase Indexer
Ao habilitar o Lily HBase Indexer, você precisa especificar a lógica de transformação do Morphlines que permitirá que esse indexador analise as atualizações da tabela de dispositivos médicos e extraia todos os campos relevantes. Vá para Serviços e escolha Lily HBase Indexer que você adicionou anteriormente. Selecione Configurações->Visualizar e Editar->Serviço-Wide->Morphlines . Copie e cole o arquivo Morphlines.
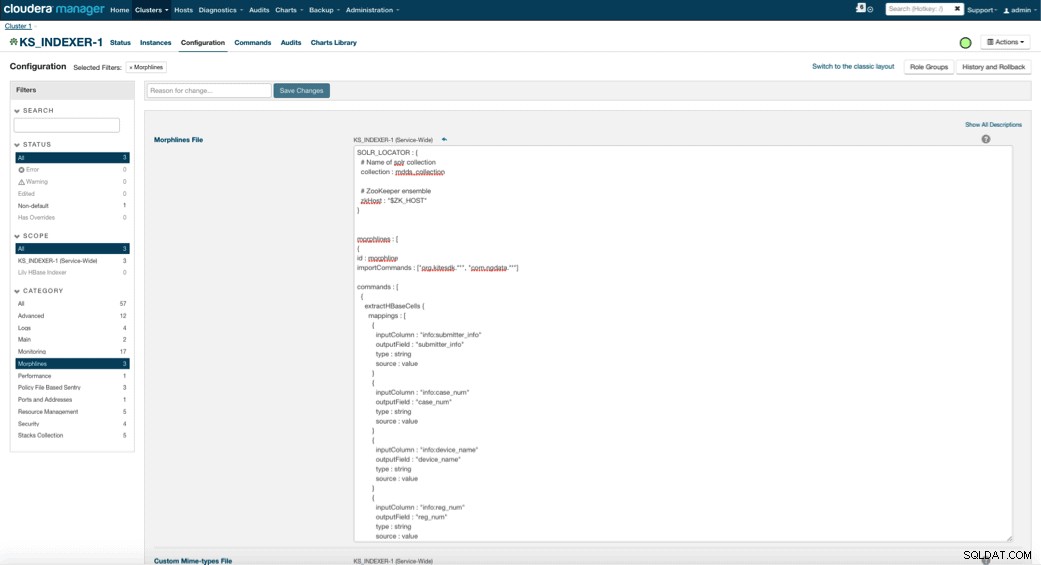
A biblioteca de morfinas de dispositivos médicos executará as seguintes ações:
- Leia os eventos de e-mail do HBase com o
extractHBaseCellscomando - Converta os carimbos de data/hora em um campo que o Solr entenda, com o
convertTimestampcomandos - Elimine todos os campos extras que não especificamos em schema.xml, com os
sanitizeUknownSolrFieldscomando
Baixe uma cópia deste arquivo Morphlines aqui.
Uma observação importante é que o campo id será gerado automaticamente pelo Lily HBase Indexer. Essa configuração é configurável no arquivo index-config.xml acima, especificando o atributo unique-key-field. É uma prática recomendada deixar o nome padrão de id—como não foi especificado no arquivo xml acima, o campo id padrão foi gerado e será uma combinação de RowID.
Acessando os dados
Você tem a opção de muitas ferramentas visuais para acessar as imagens indexadas. HUE e Solr GUI são opções muito boas. O HBase também permite várias técnicas de acesso, não apenas a partir de uma GUI, mas também por meio do shell do HBase, API e até mesmo técnicas de script simples.
A integração com o Solr oferece grande flexibilidade e também pode fornecer opções de pesquisa muito simples e avançadas para seus dados. Por exemplo, configurar o arquivo schema.xml do Solr de forma que todos os campos dentro do objeto de e-mail sejam armazenados no Solr permite que os usuários acessem corpos de mensagem completos por meio de uma pesquisa simples, com a compensação de espaço de armazenamento e complexidade de computação. Como alternativa, você pode configurar o Solr para armazenar apenas um número limitado de campos, como o id. Com esses elementos, os usuários podem pesquisar rapidamente o Solr e recuperar o rowID que, por sua vez, pode ser usado para recuperar campos individuais ou a imagem inteira do próprio HBase.
O exemplo acima armazena apenas o rowID no Solr, mas indexa todos os campos extraídos da imagem. A pesquisa do Solr neste cenário recupera IDs de linha do HBase, que você pode usar para consultar o HBase. Esse tipo de configuração é ideal para o Solr, pois mantém os custos de armazenamento baixos e aproveita ao máximo os recursos de indexação do Solr.
Exemplos de consultas
Abaixo estão alguns exemplos de consultas que podem ser feitas do aplicativo para o Solr. A ideia é que o cliente inicialmente consulte os índices do Solr, retornando o rowID do HBase. Em seguida, consulte o HBase para o restante dos campos e/ou a imagem bruta original.
- Envie-me todos os documentos que foram arquivados entre as seguintes datas:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01 -06T23:59:59.999Z PARA 2010-02-06T23:59:59.999Z]
- Forneça-me os documentos que foram arquivados sob o nome regulamentar de sistemas de raio-x móvel:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile sistema de raio-x
- Forneça todos os documentos que foram arquivados por fabricantes chineses:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Os IDs de documentos Solr são os IDs de linha no HBase; a segunda parte da consulta será para o HBase extrair os dados (incluindo o PDF bruto, se necessário).
Acesso via HUE
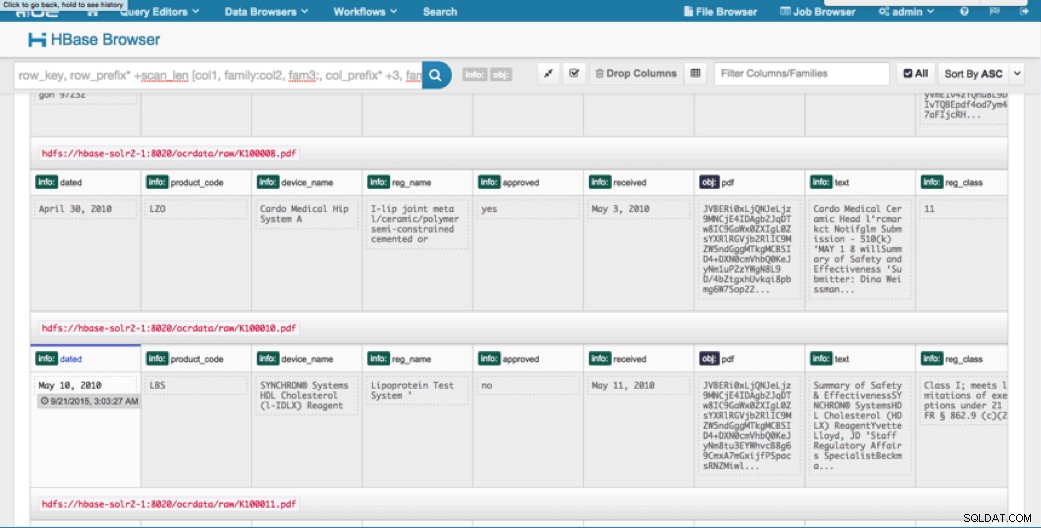
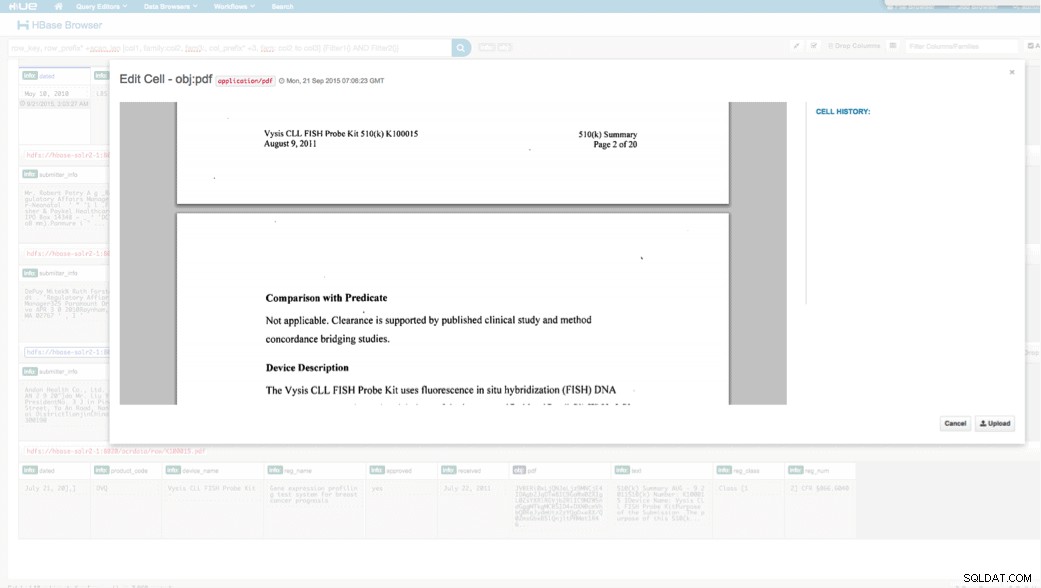
Podemos visualizar os dados enviados por meio do navegador HBase em HUE. Uma grande coisa sobre o HUE é que ele pode detectar os binários para PDF e renderizá-los quando clicado.
Abaixo está um instantâneo da visualização dos campos analisados nas linhas do HBase e também uma visualização renderizada de um dos objetos PDF armazenados como um MOB na família de colunas obj.
Conclusão
Neste post, demonstramos como usar tecnologias padrão de código aberto para executar OCR em documentos digitalizados usando um programa Spark escalável, armazenando no HBase para recuperação rápida e indexando as informações extraídas no Solr. Deve ficar claro que:
- Dado o formato de especificação da mensagem, podemos extrair campos e pares de valores e torná-los pesquisáveis via Solr.
- Esses campos de dados podem atender aos requisitos do IDMP para tornar os dados legados eletrônicos, que entrarão em vigor no próximo ano.
- Os campos e as imagens brutas podem ser persistidos no HBase e acessados por meio de APIs padrão.
Se você precisar processar documentos digitalizados e combinar os dados com várias outras fontes em sua empresa, considere usar uma combinação de Spark, HBase, Solr, juntamente com Tesseract e Leptonica. Pode poupar-lhe uma quantidade considerável de tempo e dinheiro!
Jeff Shmain é arquiteto de soluções sênior na Cloudera. Ele tem mais de 16 anos de experiência no setor financeiro com forte entendimento de negociação de segurança, risco e regulamentos. Nos últimos anos, ele trabalhou em várias implementações de casos de uso em 8 dos 10 maiores bancos de investimento do mundo.
Vartika Singh é Consultora de Soluções Sênior na Cloudera. Ela tem mais de 12 anos de experiência em aprendizado de máquina aplicado e desenvolvimento de software.